北京时间2024年7月30日17时,安徽医科大学第一附属医院曹云霞/梁丹团队联合中国科学院动物研究所郭帆团队,在《自然—细胞生物学》(Nature Cell Biology)期刊上报道了题为“Distinct dynamics of parental 5-hydroxymethylcytosine during human preimplantation development regulate early lineage gene expression”的研究论文,解析了人植入前胚胎中DNA羟甲基化的起源,命运与功能。
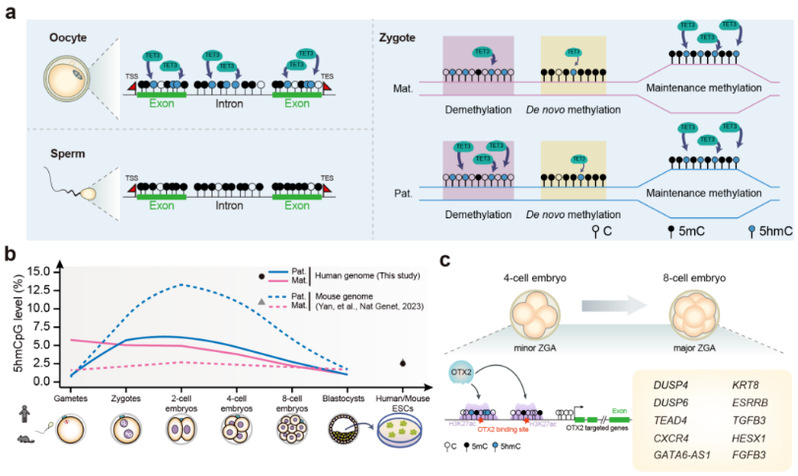
2023年郭帆研究组系统性地揭示了小鼠早期胚胎发育全时程的5hmC动态与分子调控,定量解析了Tet3和DNA复制在5hmC产生中的作用,结合小鼠遗传模型发现DNA复制和被动去甲基化途径对于5hmC的产生和基因组分布有着显著影响。除了参与DNA甲基化重编程,5hmC在小鼠早期胚胎中偏好性的富集于基因的增强子上,与基因转录调控联系紧密【7】。然而,5hmC在人早期胚胎中的动态与功能还未有研究报道,这一修饰在人-鼠间的保守性以及调控机制的异同仍不清楚,限制了我们对人胚胎发育起始阶段表观遗传机理的认识。
本研究者利用前沿的微量细胞5hmC全基因组解析方法结合胚胎成像、染色质免疫沉淀和相关功能实验,对人早期胚胎中5hmC的功能作用进行了系统解析。研究者发现人卵细胞在生长过程中经历了从头DNA羟甲基化从而累积了显著水平的5hmC;受精后,母本基因组中的5hmC继承自卵细胞,而父本基因组中的5hmC则会部分“参照”母本基因组产生,这一模式区别于小鼠早期胚胎中5hmC的不对称性分布。人早期胚胎中5hmC除了参与DNA甲基化擦除事件外,还与维持性DNA甲基化区域关联;以上结果说明人与小鼠早期胚胎中5hmC的起源与动态并不保守。人早期胚胎中部分5hmC修饰会维持至八细胞时期,并且富集在OTX2、TFAP2C和KLF4/5等转录因子的结合基序上,研究人员通过5hmC的耗竭或者异位产生实验以及人早期胚胎中OTX2的敲降实验,证明了5hmC-增强子-OTX2的互作对于人早期胚胎发育的调控作用。这些发现揭示了5hmC在人早期胚胎发育中的独特动态以及功能多面性,为理解人早期胚胎发育过程中的多能性建立、细胞谱系分化和转录-表观调控之间的关系奠定重要基础。
中国科学院动物研究所郭帆教授,安徽医科大学第一附属医院曹云霞教授、梁丹教授以及上海交通大学医学院附属第一人民医院贺小进教授为该研究论文的通讯作者。安徽医科大学第一附属医院教授梁丹、副教授纪冬梅,中国科学院动物研究所博士生燕蕊、博士后龙鑫为本文共同第一作者。安徽医科大学第一附属医院为第一作者单位。
https://doi.org/10.1038/s41556-024-01475-y
参考文献:
1. Wen, L. & Tang, F. Human Germline Cell Development: from the Perspective of Single-Cell Sequencing. Mol Cell 76, 320-328 (2019).
2. Smith, Z.D. & Meissner, A. DNA methylation: roles in mammalian development. Nat Rev Genet 14, 204-220 (2013).
3. He, Y.F. et al. Tet-mediated formation of 5-carboxylcytosine and its excision by TDG in mammalian DNA. Science 333, 1303-1307 (2011).
4. Ito, S. et al. Tet proteins can convert 5-methylcytosine to 5-formylcytosine and 5-carboxylcytosine. Science 333, 1300-1303 (2011).
5. Tahiliani, M. et al. Conversion of 5-methylcytosine to 5-hydroxymethylcytosine in mammalian DNA by MLL partner TET1. Science 324, 930-935 (2009).
6. Kriaucionis, S. & Heintz, N. The nuclear DNA base 5-hydroxymethylcytosine is present in Purkinje neurons and the brain. Science 324, 929-930 (2009).
7. Yan, R. et al. Dynamics of DNA hydroxymethylation and methylation during mouse embryonic and germline development. Nat Genet 55, 130-143 (2023).

